基因组手术三件套
我们已经知道,一个锌手指模块能够识别一个DNA 3碱基序列(见图3-9)。因为DNA一共只用到4种碱基分子,理论上存在的3碱基序列也不过是区区64种(43=64)。因此,如果我们能够找到64种不同的锌手指,分别对应一种独一无二的DNA 3碱基序列,我们就有可能通过排列组合不同数目的锌手指,实现对任意基因组DNA序列的精确定位。
比如,前面的故事里我们讲到过镰刀形红细胞贫血症,一种由于血红蛋白HBS基因第20位的A变成了T导致的单基因遗传病。如果我们用基因组精确编辑的逻辑,将错误的T再重新变成A,我们就需要一个精确的基因组GPS帮助我们迅速定位到这个错误的碱基附近,然后用一把锋利的基因组剪刀剪断错误碱基附近的DNA链,再替换上一段正确的DNA序列。
理论上,我们可以利用锌手指蛋白构造出我们想要的任何一种基因组GPS。还以镰刀形红细胞贫血症为例,假设我们希望定位的是HBS基因编码区(也就是直接编码氨基酸序列)的开始段。首先,我们可以确认前9个碱基是A-T-G-G-T-G-C-A-T。那么,只要我们能找到三个分别对应A-T-G、G-T-G和C-A-T的锌手指,再把它们串联在一起,就能人工构造出一个针对这段9碱基序列的锌手指蛋白了。当然了,对于整个人类基因组的30亿碱基对来说,9碱基序列可能还不足以标识出独一无二的位置信息。实际上,A-T-G-G-T-G-C-A-T这段序列在人类基因组里反复出现了上千次!但是不要紧,锌手指蛋白的串联特性使得我们可以通过增加一个又一个的3碱基单元,最终实现独一无二的位置标记。比如,我们发现,如果拓展到21个碱基——A-T-G-G-T-G-C-A-T-C-T-G-A-C-T-C-C-T-G-A-G——这段序列在人类基因组中就仅仅存在于HBS基因编码区的前端了。换句话说,区区7个锌手指的串联,就足够精确定位镰刀形红细胞贫血症患者体内出了问题的HBS基因!

你可能马上会问,我们真的能找到足够多的锌手指,真的可以随心所欲对它们进行组合吗?在数量方面倒是没有什么问题。在发现TFIIIA之后,科学家们在不同物种里陆续发现了上千个转录因子蛋白,它们当中有许多也和TFIIIA一样带有锌手指。很快,科学家的数据库里不同的锌手指已经积累好几百个了(当然,你可以想象对于任意一个3碱基DNA序列都存在着不只一个锌手指与之对应)。但是在做排列组合的时候,他们遇到了点不大不小的麻烦。
在上面的故事里我们曾提到,TFIIIA的9个锌手指之间是相互独立的。这也就意味着我们可以拆散天然存在的锌手指,然后装配出我们需要的组合来。换句话说,锌手指模块是“可编程的”。但在1991年人们就已经发现,一个锌手指和一段3碱基DNA序列之间并不存在完美的对应关系。利用X射线晶体学研究(同样的方法在1953年揭示了DNA双螺旋结构的秘密),美国约翰霍普金斯大学的卡尔・帕博(Carl Pabo)实验室发现,一个锌手指的立体结构要比3碱基略大,也就是说,这根手指还会向3碱基DNA的前后分别“延长”出去一小段,覆盖到前后相邻的碱基(见图3-10)。换句话说,至少从位置关系上看,前后相邻的锌手指之间并不是完全独立的,它们之间也可能存在彼此配合或干扰的关系。
举个例子,如果我们想设计一个锌手指蛋白结合一段9个碱基的DNA序列,姑且用上图出现的G-G-T-A-A-G-A-T-C这9个碱基代表吧,如果我们单独挑选出三个锌手指蛋白分别结合“G-G-T”“A-A-G”“A-T-G”,然后穿成一串,很可能是不行的。因为那根对应“G-G-T”的手指还会悄悄碰到接下来的“A”一点点,而对应“A-T-C”的手指也会偷偷去戳一下前面的“G”,这样就会干扰彼此对DNA的结合能力。因此,真要选出传说中的“黄金手指”,就需要将三根锌手指之间的关系也纳入考量。
那么我们应该怎么做呢?单纯从数字上看是很让人绝望的。我们已经说过,目前已经发现的锌手指就有成百上千个,分摊到全部64种DNA 3碱基序列上,每个序列可以分到几十个。如果锌手指可完全编程的话,挑选几个合适的锌手指再按顺序组装起来可以说是轻而易举,可选的锌手指蛋白是非常充裕的。然而,在几根手指还会互相打架的现实世界中,我们要怎么才能知道哪几根手指相处融洽,哪几根又喜欢打架呢?我们总不能把成百上千的锌手指都两个两个、三个三个,甚至四个四个地组合起来,研究确认究竟哪些才能和谐相处吧?曾经有人做过数学估计,仅仅是三个三个的排列组合来确认目前已知的所有锌手指,最终可能需要一个像整个北美洲那么大的细菌培养皿才能完成!

那怎么办呢?锌手指蛋白的命名人,1982年诺贝尔化学奖得主艾伦・克鲁格爵士用一个漂亮优雅的方法解决了这个问题。他用两个步骤完成了对三手指组合的筛选。还是拿G-G-T-A-A-G-A-T-C这段9碱基序列来说吧。他们首先筛选了能够定位G-G-T-A-A-G这段6碱基序列的锌手指组合,姑且命名为GGT-AAG(A),以及定位A-A-G-A-T-C这段6碱基序列的手指组合,姑且命名为AAG(B)-ATC。可想而知,在两个独立的筛选中,GGT锌手指和AAG(A)手指必然能够和谐相处,AAG(B)手指和ATC手指也问题不大,但是两次独立筛选获得的AAG(A)手指和AAG(B)手指虽然定位能力相同,它们自身很可能是不一样的两根手指。不过没关系,如果筛选出的手指组合数量够大,科学家们总能找到其中比较类似的A和B。这样他们就可以利用计算机模拟找出同时类似A和B的AAG(C),保证C手指能前后兼容,从而最终组装出一套好用的“黄金手指”。
看起来这个不大不小的麻烦也解决了。不过要提醒读者们注意的是,在上述筛选和计算过程的背后,是天文数字般的手指组合以及漫长的信息积累过程。哪些手指组合值得留意?到底怎么计算两根手指的相似性?这些问题筑起了高高的技术壁垒,直到今天,筛选组装出一套好用的锌手指也不是一件轻而易举的事情。请记住这一点,这对我们后面的故事很重要。
好了,锌手指的问题先说到这儿。不妨假设我们已经能够随心所欲地通过组装锌手指建造基因组GPS,定位人类基因组上的任何一段DNA序列了。那么接下来,负责剪下错误DNA序列的剪刀又在哪里呢?缝补基因组的针线呢?
1996年,在美国约翰・霍普金斯大学任教的斯里尼瓦桑・钱德拉塞格兰(Srinivasan Chandrasegaran)找到了一把很好用的基因组剪刀。说起来,钱德拉塞格兰的研究兴趣一直是基因组剪刀,只不过他从没想过自己的研究能直接应用于基因编辑工作。他的实验室一直在研究一类特殊的蛋白质分子:限制性内切酶。从名字上你们就能大概猜出,这类蛋白质的功能就是切割双链DNA分子。但是长久以来人们都知道,限制性内切酶和我们刚刚讲过的转录因子一样,都有自己偏好的DNA序列。就像TFIIIA转录因子喜欢“亲近”5S RNA基因的DNA序列,钱德拉塞格兰实验室关心的一种叫作FokI的限制性内切酶(见图3-11),也只喜欢结合一段特别的10碱基DNA序列:G-G-A-T-G-C-A-T-C-C。当然了,结合DNA之后两个蛋白质施展的手段就天差地别了:TFIIIA会启动基因的转录,而FokI则会咔嚓一刀把DNA从中切断!
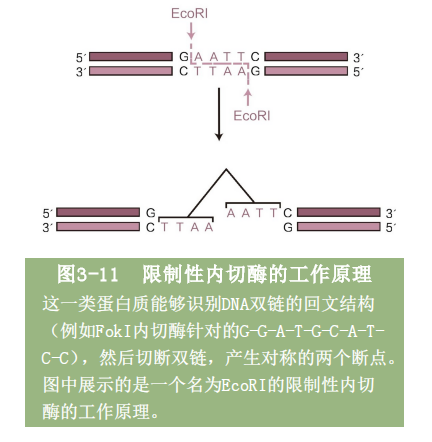
那么FokI能不能直接充当基因编辑工程里的剪刀呢?不行,原因很简单,FokI自身也具有DNA序列的特异性。换句话说,FokI其实是一个混合了基因组GPS(目标:G-G-A-T-G-C-A-T-C-C)和基因组剪刀的蛋白质。我们没办法随心所欲地指挥它去剪切任意一段我们指定的DNA序列。但在1994年,钱德拉塞格兰实验室发现,FokI蛋白里具有GPS功能的部分和具有剪刀功能的部分是截然分开的:蛋白的前半段专门负责定位,后半段专门负责切断DNA。钱德拉塞格兰和他的同事们还证明,要是把FokI的前半段替换成来自其他限制性内切酶的定位模块,就能完美地驱使FokI去切割一段完全不同的DNA序列。
这样一来事情就峰回路转了。
我们已经知道,“可编程”的锌手指蛋白是即插即用的基因组GPS。理论上,我们可以组装多个锌手指蛋白,用于定位人类基因组的任意位置。现在我们又知道,限制性内切酶FokI的后半段是把快剪刀,把它接到任何类型的DNA定位模块后面,都可以忠实执行剪切DNA的任务。那么,一个自然而然的思路就是,把锌手指蛋白和FokI的剪切模块串联在一起会发生什么。
1996年,钱德拉塞格兰终于让锌手指蛋白和FokI剪刀走到了一起,他们在FokI剪切模块的一端连上了三个不同的锌手指结构。我们已经知道,每个锌手指结构能够识别一个特定的DNA 3碱基序列。因此,如果一切真如我们所愿,这个“杂种”蛋白应该起到这样的作用:在基因组DNA上准确地找到一段特定的9碱基序列,然后在那里剪开DNA长链。实验取得了完美成功,而这个创造历史的“杂种”蛋白,被钱德拉塞格兰命名为“锌手指核酸酶”(见图3-12),正式进入了基因编辑的工具箱。
有了GPS,有了剪刀,再找到基因组针线,我们是不是就可以动手修复遗传病的基因缺陷了?实际情况比我们的计划还要更美好一点,基因组DNA有天然的针线可以用,根本用不着我们操心。更妙的是,细胞里居然有两套完全不同的基因组针线,一套可以帮助我们破坏一个原本正常的基因,一套可以帮助我们修复错误的基因!
这里说的基因组针线,有一个更专业的名字——“DNA修复机制”。当然了,和我们故事里讲过的基因组GPS锌手指蛋白、基因组剪刀FokI一样,DNA修复机制在细胞里已经存在了亿万年,自然也不是专门为我们给基因动手术的任务准备的。它的首要使命是保证基因组DNA的完整性,保证哪怕历经严酷的环境挑战,生命的遗传物质都不会被轻易破坏。
在第一章的故事里,大家应该已经完全理解遗传物质DNA对生命现象的极端重要性了。而从第2章的故事里,你们应该也明白,那些DNA分子上哪怕是极其微小的差错,都有可能引起致命的人类疾病。(想想镰刀形红细胞贫血症,仅仅是由于某个单一碱基的错误!)但我们还需要知道的是,想要维持基因组DNA的完整性是个极端困难的任务。就拿人体来说吧,从一个受精卵开始,经过数十万亿次的细胞分裂,受精卵细胞中的DNA分子经历数十万亿次的半保留复制,才造就了你我今天的模样。在此过程中,我们身体里的DNA分子总长度已经从几米扩展到了上千亿千米,足够从地球出发往返冥王星好几百次!要保证这几十万亿次的DNA复制不出差错,保证DNA分子在各种高能射线和化学毒物的持续攻击下不出问题,我们的细胞进化出了各种各样的DNA修复机制,可以敏锐地发现刚刚露苗头的任何错误,然后第一时间把错误碱基替换掉。也多亏了这些DNA修复机制,DNA复制的错误率低得惊人——10-9(每复制十亿碱基可能会出错一次)。
DNA分子面临的最大威胁可能就是彻底断裂了!想象一下,一条拉直来看长达数米的DNA长链一旦从中突然断裂,如何在细胞核里重新找到两个断点,又怎么重新准确地连接呢?考虑到DNA链条和细胞核的直径相差近1 000倍,细胞核里还拥挤地穿梭着各种各样的分子,这个任务的难度堪比从一个装满各色海洋球的游乐场里,找出仅有的两个标记着星形图案的红色海洋球……
所幸,细胞里早就为我们准备好了应对这种灾难性事件的预案。在20世纪90年代初,人们就已经发现,如果DNA双链真的出现了断裂,细胞会利用两种方法进行紧急修复(见图3-13)。其中一个办法很直接:找到两个DNA断点,不管三七二十一直接粘上就行。这样的好处是简单快捷,坏处是容易出错。试想一下,如果断头的DNA不小心丢掉或增加了好几个碱基(这是很可能发生的),直接粘上不就永久性地失去或增加了那几个碱基的信息吗?另一个方法则要小心一些:找到断点之后不是直接粘上,而是先在附近找找有没有序列相似的DNA分子,以它为模板进行修复。这样万一真的出现了不该有的丢失或增加,也可以及时改正。当然了,这种看起来更可靠的方法也有问题:必须能在附近找得到序列相似的DNA才行。这个条件一般只有在细胞分裂过程中才能满足。那时候DNA已经复制完毕,但细胞尚未完成分裂,所以一个细胞里就出现了两套完全一样的DNA分子。如果其中一套出现了断裂,自然就可以利用另一套作为修复模板了。
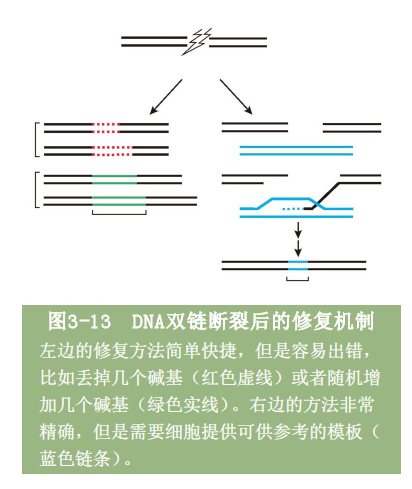
在这里唠叨了这么多,其实就是想要说明当锌手指核酸酶在基因组中找到合适的位置并进行剪切之后,细胞自身就可以完成缝缝补补的针线活。其中存在两种不同的缝补办法。如果仅仅对基因组DNA一剪了之,没有现成模板可利用的细胞只能用前一种简单粗暴的办法进行修复,难免会出现不该有的序列错误。如果在剪切的同时主动给细胞提供一段可供参考的模板序列,细胞就会启动后一种修复机制,按照模板的序列信息老老实实地修复断裂的DNA。
我们马上可以想到,这两种缝补办法其实各有各的用处!如果我们想破坏掉一个正常基因的功能,就像破坏掉正常人体内的CCR5基因以期阻止HIV的入侵,前一种不够精确的缝补方法就可以帮助我们达到目的。如果我们想要修复一个原本就有问题的基因,就像镰刀形红细胞贫血症患者体内的HBS基因,那么后一种缝补方法正好能满足需求。
讲到这里,我们已经找到了精确基因编辑所需的工具三件套:
●基因组GPS:锌手指蛋白组合;
●基因组剪刀:FokI蛋白的剪切模块;
●基因组针线:细胞内天然存在的两套DNA断点修复机制;
在20世纪的尾巴上,基因编辑已经万事俱备。而在近20年过去后的今天,基因编辑又走到哪一步了呢?它是否已经走出实验室,走进医院和病房了呢?它是不是已经改变了万千病人的命运呢?
遗憾地说,并没有。




