-
1.1前 言
-
1.2细 胞
-
1.2.1细胞的结构
-
1.2.2细胞中的化学成分
-
1.3基因与基因组
-
1.3.1什么是基因
-
1.3.2基因的组成
-
1.3.3核 酸
-
1.3.4基因的位置
-
1.3.5基因的一般特性
-
1.3.6基因分类
-
1.3.7基因组
-
1.4染色体
-
1.4.1什么是染色体
-
1.4.2染色体的结构变异
-
1.4.3染色体的数目变异
-
1.4.4染色体与男女性别决定
-
1.5DNA
-
1.5.1什么是DNA
-
1.5.2DNA的结构与组成
-
1.5.3DNA的功能
-
1.5.4RNA的功能
-
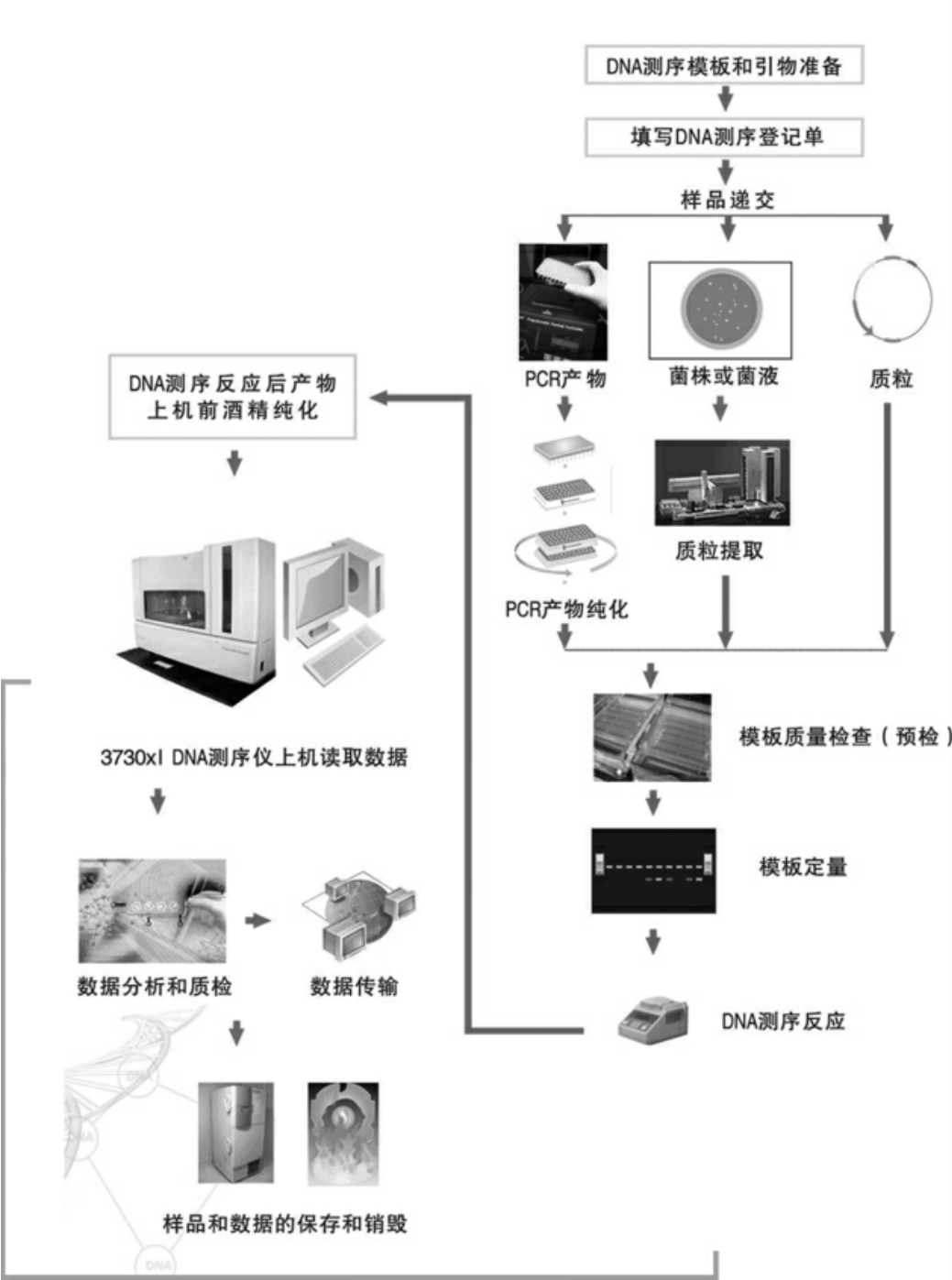
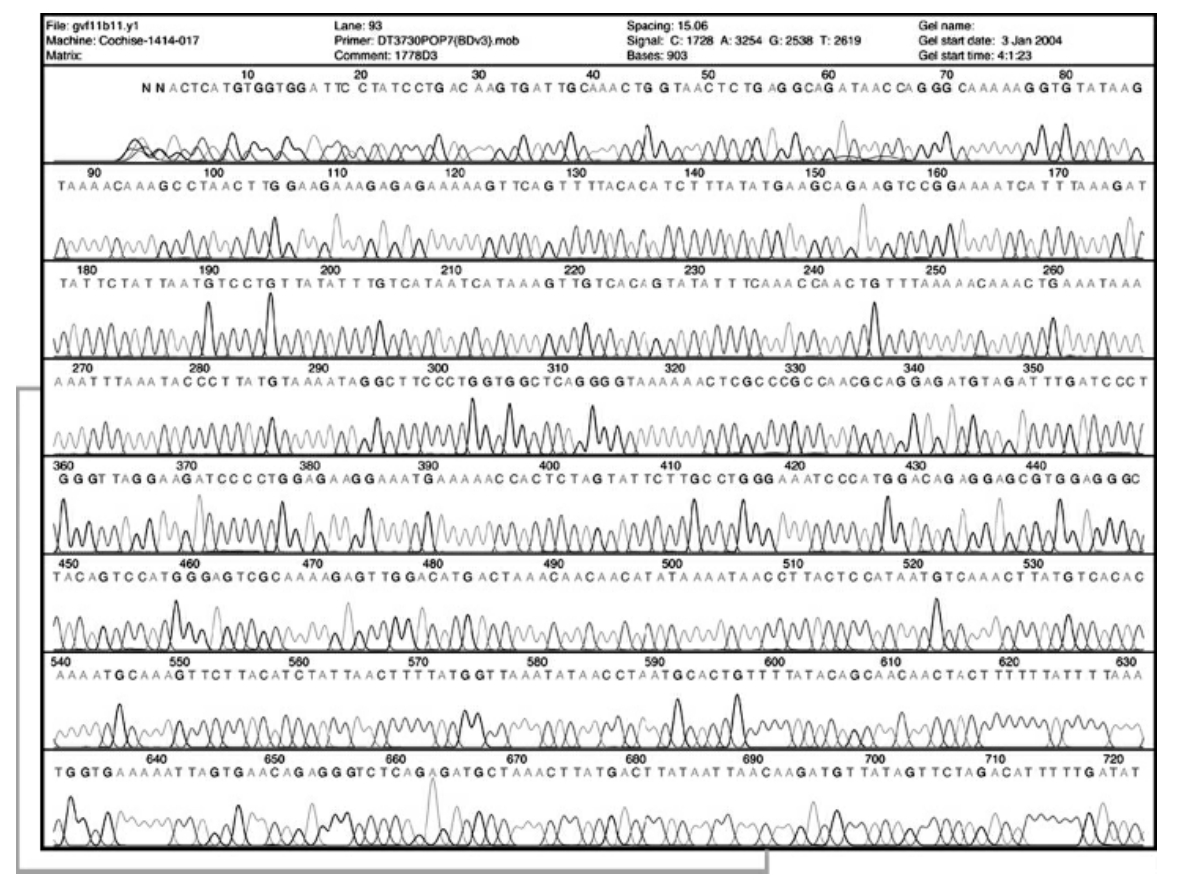
1.5.5DNA测序
-
1.5.6DNA与遗传的关系
-
1.5.7DNA指纹探秘
-
1.6基因与生命遗传
-
1.6.1遗传的分离定律
-
1.6.2遗传的自由组合定律
-
1.6.3遗传的连锁与互换规律
-
1.6.4基因是怎样控制遗传的
-
1.6.5性格形成源于基因
-
1.6.6基因突变
-
1.7基因与疾病
-
1.7.1基因与遗传性疾病
-
1.7.2基因与遗传易感性疾病
-
1.8基因工程
-
1.8.1基因工程的概念
-
1.8.2基因工程的出现和创立
-
1.8.3基因工程是怎样“施工”的
-
1.8.4基因的诊断技术
-
1.8.5基因医病
-
1.8.6基因工程疫苗
-
1.8.7转基因食品
-
1.8.8基因农业
-
1.8.9转基因动物
-
1.8.10动物制药厂
-
1.8.11细菌制药厂
-
1.8.12人造基因血液
-
1.8.13DNA的“分子手术”
-
1.8.14生物芯片
-
1.8.15基因武器
-
1.9基因克隆
-
1.9.1什么是基因克隆
-
1.9.2基因克隆的秘密
-
1.9.3首例克隆“多莉”羊
-
1.9.4单亲雌核生殖
-
1.9.5微生物克隆技术
-
1.9.6植物克隆技术
-
1.9.7动物克隆技术
-
1.9.8克隆人类
-
1.9.9我国的克隆成果
-
1.10人类基因组计划
-
1.10.1人类基因组计划的含义
-
1.10.2人体基因的重大发现
-
1.10.3人类基因组计划的实施
-
1.10.4后基因组计划
-
1.10.5蛋白质组学计划
-
1.11我国加盟人类基因组计划
-
1.11.1加盟世界基因组织
-
1.11.2生物资源基因组计划
-
1.11.3人类功能基因研究
-
1.11.4后基因时代的中国战略
1
破解基因密码